
Bioinformática: De las computadoras al genoma
La bioinformática es un área interdisciplinaria que incluye herramientas computacionales utilizadas en la biología.
Por sorprendente que parezca, se aplica principalmente en el área de la biología molecular, a través de métodos analíticos y matemáticos que se unen con la simulación informática, para analizar e interpretar una extensa colección de datos biológicos, como las secuencias genéticas, poblaciones celulares y secuencias proteicas para conocer y hacer nuevas predicciones genéticas
El término “bioinformática” tiene su origen en la década de los años ‘70s. En ese entonces, se asociaba directamente con el uso de herramientas computacionales para analizar datos biológicos que se obtenían a nivel experimental.
Hoy en día sigue la misma dirección, pero con la implementación de nuevas técnicas biológicas, software y programas computacionales más sofisticados que permiten llegar a comprender fenómenos biológicos que hace un par de décadas era casi imposible de integrar.
Un ejemplo concreto, son las técnicas de secuenciación masiva que se han empleado en la actualidad para determinar la composición genética de las variantes del virus SARS-Cov2. De no ser por ellas no podríamos saber los genes y mecanismos involucrados en la infección, permitiéndonos de esta manera conocer las proteínas que posee el virus en su estructura o bien, las proteínas que utiliza para infectar o para replicarse dentro de las células hospedadoras.
Lo anterior posiciona a la bioinformática como una rama sólida e indispensable en la ciencia, pues, a través de ella, es posible entender procesos cinéticos, moleculares o bien descubrir y clasificar nuevos microorganismos y/o especies biológicas mediante el análisis de su composición genómica y proteica.
Sin embargo, muchos se preguntan: ¿cómo es posible saber toda esa información? La respuesta es, por medio de distintos programas diseñados con algoritmos matemáticos y analíticos específicos que permiten obtener datos que revelen el orden o comportamiento del objeto que se estudia.
Existe una amplia variedad de programas bioinformáticos que se utilizan en la ciencia básica y aplicada. Entre estos destacan los que funcionan como archivadora, vale decir, que almacenan y organizan datos de secuencias de ácidos nucleicos (ADN o ARN) o de secuencias proteicas.
Actualmente hay numerosas bases de datos biológicas disponibles en la web, siendo la base de datos Protein Data Bank (Img. 1) una de las más populares y antiguas. En esta se encuentra depositada la información de estructuras tridimensionales de biomoléculas como proteínas y ácidos nucleicos, determinadas mediante cristalografía de rayos X, permitiendo acceder y estudiarlas con una finalidad terapéutica o farmacológica.
Asimismo, la base de datos de ácidos nucleicos GenBank (Img. 1) se ha convertido en una de las herramientas bioinformáticas más completa, la cual contiene anotaciones de secuencias nucleotídicas de una gran cantidad de organismos. Esta plataforma cuenta con secuencias de ADN, ARN mensajero (ARNm) y ADN complementario (ADNc).

Imagen 1. Base de datos Protein Data Bank a la derecha. Es considerada una de las más antiguas y populares en su tipo. A la izquierda tenemos a GenBank, que es la más completa con anotaciones de secuencias nucleotídicas de una gran cantidad de organismos.
Además de las bases de datos bioinformáticas, existen numerosos modelos computacionales que permiten comparar y analizar secuencias nucleotídicas o proteicas, identificando así, los patrones de similitud entre ellos.
Por ejemplo, el grado de similitud entre dos proteínas a partir del análisis de las regiones conservadas o variables de las proteínas. En base a esta comparación, se puede construir árboles filogenéticos o proteómicos (Img. 2) para determinar las relaciones evolutivas entre las especies u organismos que se estén estudiando.
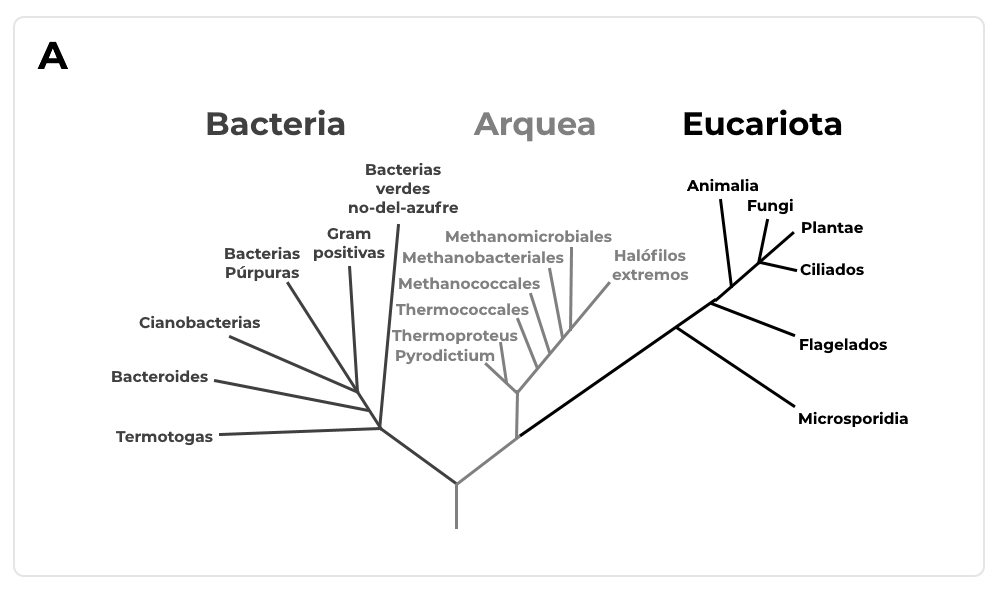
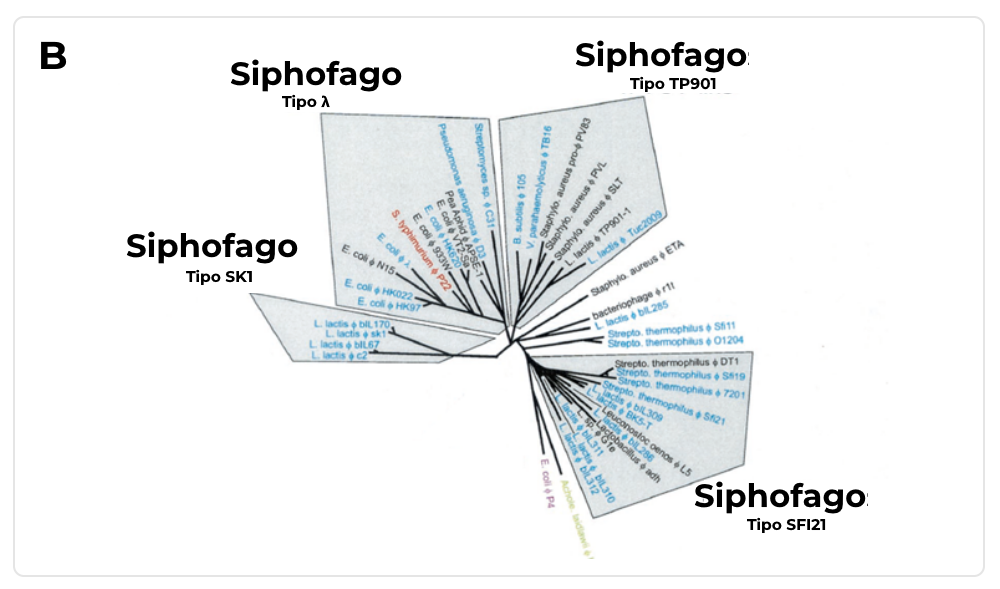
Imagen 2. En el panel A se aprecia un árbol filogenético que muestra relaciones evolutivas entre distintos dominios. En el panel B se observa un árbol proteómico de Siphofagos. El árbol se construyó desde 45 genomas secuenciados de fagos y compara dichas secuencias con programas bioinformáticos como alineamiento de secuencias BlastP y otros.
Ante el progresivo avance de la bioinformática, universidades en todo el mundo han creado carreras de pregrado y programas de postgrado enfocados en formar profesionales competentes en esta área para que sean capaces de usar los lenguajes de programación y herramientas informáticas en el análisis de datos biológicos. También para diseñar software y programas que sean aplicados en la biología, pudiendo crear nuevas funciones de análisis y/o mejorar las ya existentes.
En este artículo, hemos visto que el desarrollo y la inclusión de nuevas tecnologías como la bioinformática han beneficiado con creces a la ciencia, pues ha dado paso a la creación de nuevas herramientas que nos ofrecen plataformas eficientes para identificar desde nuevas biomoléculas a nuevas entidades biológicas.
